Comprendre les maladies génétiques rares avec l'expansion du code génétique
- Recherche
le 3 avril 2024

Une étude publiée par l'équipe de Michèle Studer à l'Institut de Biologie Valrose dans la revue Protein Science en mars 2024, propose une nouvelle approche pour comprendre les bases moléculaires des maladies génétiques, en reliant les mutations spécifiques du gène NR2F1 aux symptômes variés des patients atteints du BBSOAS. Ces avancées pourraient également avoir des applications importantes pour l'étude d'autres maladies génétiques.
Pour améliorer la précision des diagnostics et des traitements, il est crucial de comprendre la relation entre les mutations génétiques et les symptômes des maladies. Cela nécessite notamment une compréhension approfondie de la manière dont les mutations affectent la structure et la fonction des protéines.
Dans cette étude, les chercheurs ont utilisé une approche combinant des simulations informatiques de la structure protéique de différents mutants génétiques de NR2F1 avec des expériences cellulaires pour étudier les effets des mutations sur la prolifération et la survie des cellules. Ils ont également employé une technologie innovante de biologie synthétique appelée « expansion du code génétique » (GCE) pour introduire des acides aminés non conventionnels dans la protéine NR2F1. En utilisant un acide aminé dit « photoréticulant », cad capable de former des liaisons covalentes avec des molécules voisines lorsqu'il est exposé à la lumière UV, les chercheurs ont ainsi pu visualiser les interactions protéiques de NR2F1 et de ses différents mutants avec une précision inégalée. Cette approche a révélé comment NR2F1 interagit avec d'autres protéines et comment ces interactions peuvent être perturbées par les mutations génétiques. De plus, cette méthode a identifié un nouveau partenaire protéique de NR2F1, CRABP2, qui joue un rôle dans sa localisation cellulaire en réponse à des signaux spécifiques.
En résumé, cette étude propose une nouvelle approche pour comprendre les bases moléculaires des maladies génétiques, en reliant les mutations spécifiques du gène NR2F1 aux symptômes variés des patients atteints du BBSOAS. Ces avancées pourraient également avoir des applications importantes pour l'étude d'autres maladies génétiques.
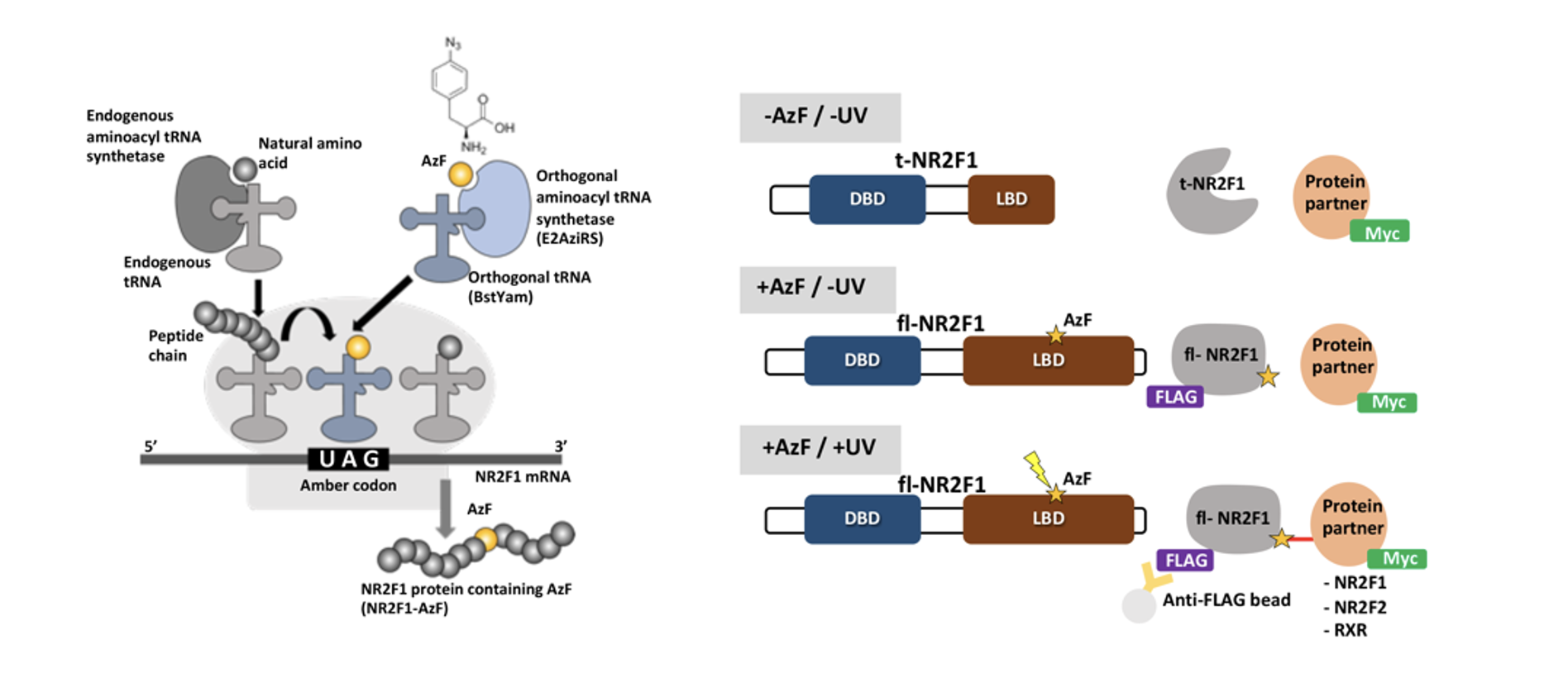
A gauche, principes de l'incorporation spécifique d'un site d'un acide aminé photoréticulant par suppression du codon de l'ambre. L'aminoacyl-ARNt synthétase endogène charge l'ARNt endogène avec l'acide aminé qu'il reconnaît. L'aminoacyl-ARNt entre dans le ribosome et ajoute l'acide aminé au codon correspondant. Pour l'incorporation de l'AzF dans NR2F1, l'aminoacyl-ARNt synthétase orthogonale (E2AziRS) catalyse l'aminoacylation entre l'AzF et l'ARNt orthogonal (BstYam). L'ARNt chargé d'AzF entre dans le ribosome et incorpore l'AzF en réponse au codon ambre désigné. La traduction se poursuit et produit la protéine NR2F1 contenant l'AzF. A droite, diagramme montrant la réticulation spécifique aux différents sites de la protéine NR2F1, induite par les UV via l'AzF. NR2F1 est produit sous forme tronquée (t-NR2F1) en l'absence d'AzF en raison du codon ambre désigné. En présence d'AzF, la suppression du codon ambre permet l'incorporation d'AzF à la position sélectionnée du codon ambre et la protéine NR2F1 portant AzF est traduite en une forme complète marquée par FLAG (fl-NR2F1). Sous irradiation UV (365 nm), AzF forme une liaison covalente entre FLAG-NR2F1 et des partenaires putatifs marqués par Myc (NR2F1, NR2F2, RXRα). Les protéines dans le complexe réticulé peuvent être co-immunoprécipités avec NR2F1 en utilisant des billes anti-FLAG conjuguées à des anticorps et détectées dans un complexe de poids moléculaire plus élevé par immunoblot. Pour plus de détails, voir Marino et al., Protein Sci. 2024.
- Pour en savoir plus
-
https://onlinelibrary.wiley.com/doi/10.1002/pro.4953
Marino V, Phromkrasae W, Bertacchi M, Cassini P, Chakrabandhu K, Dell'Orco D, Studer M.
“Disrupted protein interaction dynamics in a genetic neurodevelopmental disorder revealed by structural bioinformatics and genetic code expansion”. Protein Sci. 2024 Apr;33(4):e4953. doi: 10.1002/pro.4953. - Contacts auteurs :
-
Michèle Studer | Directrice de recherche INSERM | +33 489150720 | Michele.studer@univ-cotedazur.fr




