Cibler la protéase majeure du SARS-CoV-2 pour fabriquer un médicament efficace contre ce coronavirus
- Recherche
Mi-juin 2020, on comptait plus de 7,7 millions de cas d’infection confirmée par ce virus, dont plus de 427 000 ayant entraîné le décès, ce qui en fait la pandémie la plus grave de ces dernières décennies. Comme il n’existe à ce jour aucun médicament pour soigner cette infection, des essais cliniques sont en cours pour évaluer l’efficacité de différents médicaments disponibles sur le marché, dont certains sont utilisés pour traiter le Sida (syndrome d’immunodéficience acquise), dû à une infection chronique par le VIH (virus de l’immunodéficience humain). Quels sont ces derniers médicaments et comment agissent-ils pour limiter l’infection ?
1 Université Côte d’Azur, Institut de biologie Valrose, Faculté de médecine, 28 avenue Valombrose, 06107 Nice, France.
2 Université Côte d’Azur, Institut de chimie de Nice, Faculté des sciences, 28 avenue Valrose, 06108 Nice, France.
Nous sommes de plus en plus souvent confrontés à des maladies infectieuses à l’origine d’épidémies, pour lesquelles il n’existe pas encore de traitement efficace. La plupart de ces épidémies sont dues à l’émergence de microorganismes pathogènes, notamment des virus, qui se transmettent naturellement d’animaux vertébrés aux humains. L’ampleur de l’épidémie va ensuite dépendre de la capacité de transmission du pathogène entre individus contaminés.
En décembre 2019, un nouveau coronavirus, SARS-CoV-2, a causé un syndrome respiratoire aigu sévère (SRAS) chez de nombreux habitants de la province du Hubei en Chine, et, à la faveur des voyages intercontinentaux, s’est rapidement répandu dans le monde entier [1]. Mi-juin 2020, on comptait plus de 7,7 millions de cas d’infection confirmée par ce virus, dont plus de 427 000 ayant entraîné le décès, ce qui en fait la pandémie la plus grave de ces dernières décennies. Comme il n’existe à ce jour aucun médicament pour soigner cette infection, des essais cliniques sont en cours pour évaluer l’efficacité de différents médicaments disponibles sur le marché, dont certains sont utilisés pour traiter le Sida (syndrome d’immunodéficience acquise), dû à une infection chronique par le VIH (virus de l’immunodéficience humain). Quels sont ces derniers médicaments et comment agissent-ils pour limiter l’infection ?
Les coronavirus et leur propagation
Les coronavirus provoquent généralement des infections bénignes des voies respiratoires, comme une rhinite. Cependant, en 2003, un coronavirus dénommé SARS-CoV-1 a été reconnu responsable d’un SRAS ayant causé la mort de plus de 750 personnes dans le monde [2].
(→) Voir le Repères d’I. Tratner, m/s n° 8-9, août-septembre2003, page 885
Les génomes du SARS-CoV-1 et du SARS-CoV-2 présentent 79 % d’identité dans leurs séquences nucléotidiques, et les protéines virales synthétisées ont donc, elles aussi, des structures très semblables [3]. Le génome des coronavirus est composé d’un simple brin d’acide ribonucléique (ARN) portant l’information pour produire plusieurs protéines qui permettent au virus de se multiplier dans la cellule infectée et de créer de nouvelles particules virales infectieuses. La première protéine produite est une réplicase virale (ARN polymérase ARN-dépendante), une enzyme qui fabrique des nouvelles molécules d’ARN en prenant comme modèle le brin d’ARN viral. Ces ARN nouvellement synthétisés sont ensuite traduits par les ribosomes et enzymes de la cellule infectée pour produire les protéines qui formeront les nouvelles particules virales (virions). Une longue poly-protéine unique est d’abord synthétisée. Elle sera ensuite coupée par l’une des deux protéases virales issues de cette poly-protéine par auto-clivage [18] (→) en protéines de tailles plus petites, qui formeront notamment la capside virale, c’est-à-dire la coque dans laquelle l’ARN viral sera « empaqueté ». C’est ainsi que se forment les nouveaux virus infectieux.
(→) Voir la Synthèse de D. Juckel et al., page 633 de ce numéro
Plusieurs étapes sont donc nécessaires pour la formation de nouvelles particules virales : entrée du virus dans la cellule qu’il va infecter, copie de l’ARN viral, synthèse de la poly-protéine et sa découpe en protéines fonctionnelles de tailles plus petites, et enfin, reconstitution de nouvelles particules virales infectieuses et leur relargage à l’extérieur de la cellule infectée (Figure 1).
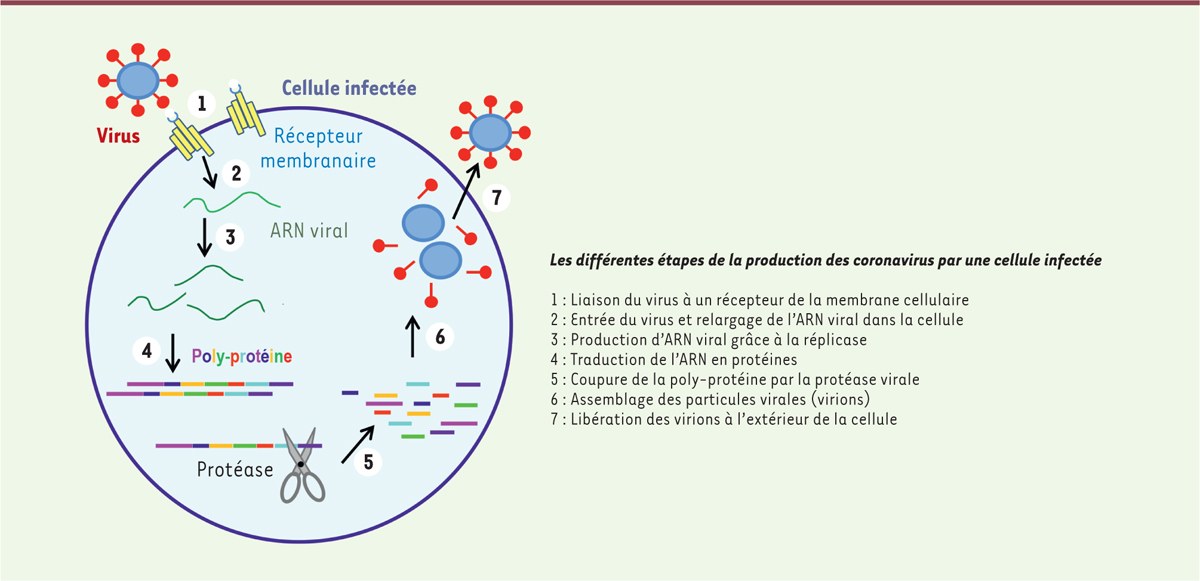
Comment peut-on limiter l’infection par les coronavirus ?
Plusieurs stratégies existent pour lutter contre les virus :
1) On peut envisager de bloquer l’entrée du virus dans la cellule si on connaît le « récepteur » de la membrane plasmique sur lequel il se fixe, ce qui n’est pas toujours le cas. Parfois, cette stratégie s’avère impossible à mettre en œuvre car le récepteur en question est nécessaire à d’autres fonctions cellulaires vitales.
2) On peut tenter d’empêcher la formation d’ARN viral.
3) On peut aussi envisager de bloquer la protéase du virus pour l’empêcher de couper la poly-protéine virale synthétisée par la cellule infectée. Les particules virales ne pourront ainsi pas s’assembler dans la cellule, ce qui stoppera l’infection. Cette stratégie a été utilisée avec succès pour traiter plusieurs infections virales dans des maladies comme le Sida ou l’hépatite C, bien que cela ne permette pas d’éliminer complètement le virus de l’organisme hôte.
Les protéases virales, des cibles de choix pour fabriquer des médicaments antiviraux
Les protéases virales sont essentielles à la production de virions par la cellule infectée car elles participent à une étape de maturation en coupant les poly-protéines virales synthétisées à des sites bien précis. Elles sont spécifiques d’un virus donné et agissent sur les protéines virales, ainsi que sur quelques protéines de la cellule hôte, pour favoriser la réplication virale [4]. C’est pour cela qu’elles suscitent un grand intérêt pour stopper la propagation des épidémies virales.
Le premier inhibiteur de la protéase (IP) du VIH, le saquinavir, a été mis sur le marché en 1995 pour traiter les malades atteints du Sida [5]. Depuis, d’autres inhibiteurs de cette protéase ont été développés pour lutter contre la maladie, comme le lopinavir [6] qui, selon l’Organisation mondiale de la santé (OMS), est un des médicaments essentiels pour traiter le Sida. Ces médicaments ciblant la protéase virale, souvent prescrits en association avec ceux qui ciblent la réplicase, ont permis de réduire la charge virale des individus atteints de la maladie.
Cependant les IP dirigés contre la protéase du VIH ont des effets indésirables, dont les mécanismes sont étudiés par notre laboratoire depuis plus de dix ans. Ils induisent notamment une redistribution marquée du tissu adipeux ou lipodystrophie, préjudiciable : le tissu adipeux sous-cutané s’atrophie et la masse grasse viscérale augmente. De plus, les lipodystrophies vont de pair avec des altérations métaboliques comme une hyperlipidémie ou une résistance à l’insuline, qui sont à l’origine de maladies cardio-vasculaires [7, 8] (→).
(→) Voir la Synthèse de J. Capeau et al., m/s n° 5, mai 2006, page 531
Nous avons montré que les IP empêchaient la formation d’adipocytes, qu’ils modifiaient les précurseurs adipocytaires ainsi que le métabolisme des adipocytes [9-11], et nous avons caractérisé le mécanisme par lequel ils induisent une résistance à l’insuline [12].
Identification de la protéase majeure du SARS-Cov-2
Pour comprendre le fonctionnement des protéases virales, il faut certes connaître leur séquence d’acides aminés, mais aussi leur structure tri-dimensionnelle. Cela permet alors de caractériser précisément le site catalytique de l’enzyme, qui sera la cible des molécules à usage thérapeutique capables de le bloquer. Les coronavirus possèdent deux protéases [13]. La structure cristalline de la protéase majeure du SARS-CoV-2, 3CLpro (3C-like proteinase), vient d’être publiée [14]. Sa séquence protéique est identique à 96 % à celle des protéases 3CLpro des autres coronavirus, qui ont été très étudiées [15, 18] (→). Elle coupe la poly-protéine virale 11 fois au niveau de motifs Leu-Gln-(Ser/Ala/Gly). La deuxième protéase du SARS-CoV-2, PLpro (papain-like protease), a des propriétés différentes de celles de 3CLpro, notamment une activité déubiquitinase, et elle pourrait intervenir pour altérer la réponse immunitaire innée de l’hôte infecté. La structure cristalline de cette protéase n’est pas encore connue.(→) Voir la Synthèse de D. Juckel et al., page 633de ce numéro
Le principe d’action des molécules bloquant les protéases est schématisé dans la Figure 2 . Des inhibiteurs de la protéase 3CLpro des coronavirus déjà connus ont été testés pour leur capacité à inhiber celle du SARS-CoV-2. Une molécule de la famille des α-kétoamides a été retenue pour son efficacité d’insertion dans le site catalytique de la protéase, ce qui bloque son activité et permet de réduire considérablement la production virale dans des cellules de poumon humain infectées avec le SARS-CoV-2 in vitro, ainsi que dans des modèles d’études précliniques [14]. La tolérance de cette molécule par l’organisme, la voie d’administration la plus adaptée, et ses effets indésirables devront être évalués avant de pouvoir éventuellement l’utiliser pour traiter les patients atteints du COVID-19.
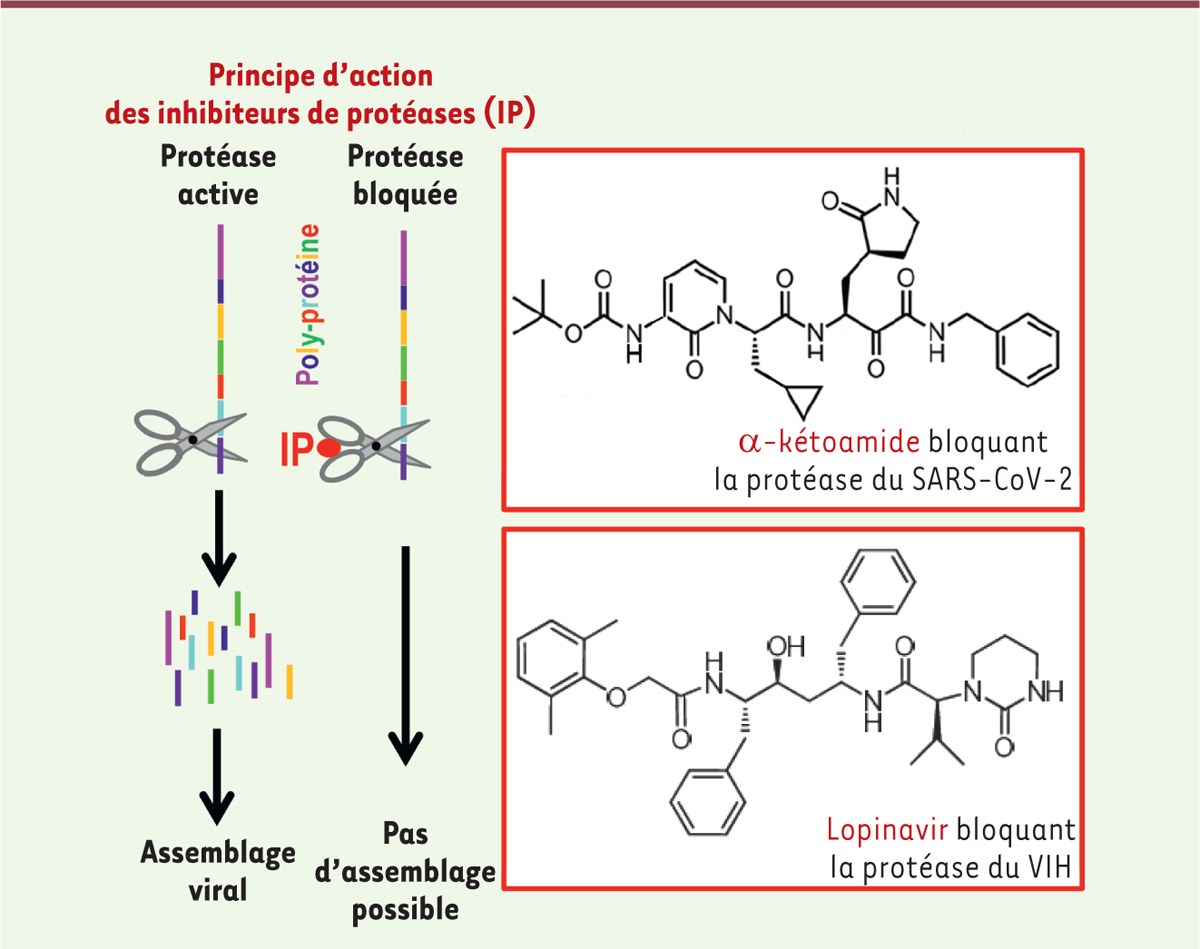
Les médicaments ciblant la protéase du VIH peuvent-ils bloquer la protéase 3CLpro du SARS-CoV-2 ?
Comme il n’y a pas encore de médicament contre le SARS-CoV-2, des essais cliniques pour tester l’efficacité des inhibiteurs de la protéase du VIH sur le COVID-19 ont été menés en Chine [16], et d’autres sont en cours en Europe. Le VIH n’est pas apparenté aux coronavirus, et la réplication de son matériel génétique s’effectue par un mécanisme différent. Mais il produit lui aussi une poly-protéine qui doit être coupée 9 fois par une protéase pour former les nouvelles particules virales. Cette protéase n’appartient pas à la même famille que celle du SARS-CoV-2 puisqu’il s’agit d’une protéase aspartique, comme les pepsines, les cathepsines, et les rénines, qui sont présentes naturellement dans les cellules eucaryotes [17]. Pour la protéase du VIH également, les données de la cristallisation ont grandement facilité la conception d’IP efficaces. Aujourd’hui, il en existe dix approuvés par la Food and drug administration aux États-Unis, dont le mélange lopinavir-ritonavir qui a été testé chez les malades atteints du COVID-19. L’essai clinique chinois a inclus 199 patients, dont 99 ont été traités par les IP du VIH tandis que 100 ont reçu des soins standards. Les résultats indiquent que les IP du VIH n’apportent pas de bénéfice pour traiter les patients atteints de COVID-19. Leurs effets indésirables ont même conduit à arrêter prématurément le traitement chez 13 patients [16]. Ce résultat, bien que décevant, est en accord avec les différences de structure et de fonctionnement entre les protéases du VIH et du SARS-CoV-2. Dans l’essai clinique adaptatif européen Discovery coordonné par l’Inserm et dans l’essai international Solidarity initié par l’OMS, le lopinavir et le ritonavir, associés ou non à l’interféron b-1a, ont été inclus en plus des soins standards, pour évaluer leurs effets contre le SARS-CoV-2. Les résultats devraient apporter un éclairage supplémentaire sur la pertinence d’utilisation des IP du VIH pour lutter contre des infections virales dues à un virus différent.
En conclusion
La stratégie la plus rapide pour tenter de trouver des médicaments efficaces contre le COVID-19 est de mener des essais cliniques avec des médicaments déjà utilisés contre d’autres maladies, puisque l’on sait comment les administrer et qu’on en connaît les doses efficaces (stratégie du « repositionnement de médicament », ou drug repositioning en anglais). Cependant, les inhibiteurs d’une protéase donnée ne sont pas nécessairement efficaces pour bloquer une autre protéase, qui a des propriétés structurales et fonctionnelles différentes. Des molécules de la famille des α-kétoamides, capables de bloquer la protéase 3CLpro du SARS-CoV-2, représentent un espoir réel pour traiter les patients. Certaines ont déjà été utilisées en clinique, comme par exemple le Telaprevir ou le Boceprevir, qui furent parmi les premiers inhibiteurs de protéase prescrits dès 2011 pour traiter les patients atteints d’hépatite C. Le développement d’inhibiteurs de protéase devrait bénéficier de moyens conséquents pour pouvoir disposer rapidement d’un traitement efficace contre le COVID-19.
- Contact chercheur
-
Contact chercheur : Annie Ladoux : ladoux@unice.fr
https://www.medecinesciences.org/en/articles/medsci/full_html/2020/06/msc200106/msc200106.html#R1 - Références
-
1 Coronaviridae study group of the international committee on taxonomy of V. The species severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol 2020; 5 : 536–44. [Google Scholar]
2 Tratner I. SRAS : 1. Le virus. Med Sci (Paris) 2003 ; 19 : 885–891. [Google Scholar]
3 Lee EYP, Ng MY, Khong PL. COVID-19 pneumonia: what has CT taught us? Lancet Infect Dis 2020; 20 : 384–5.[Google Scholar]
4 Jager S, Cimermancic P, Gulbahce N, et al. Global landscape of HIV-human protein complexes. Nature 2011 ; 481 : 365–370. [Google Scholar]
5 James JS. Saquinavir (Invirase): first protease inhibitor approved–reimbursement, information hotline numbers. AIDS Treatment News 1995 ; 1–2. [Google Scholar]
6 Hurst M, Faulds D. Lopinavir. Drugs 2000; 60 : 1371–9; discussion 80–1. [Google Scholar]
7 Capeau J, Caron M, Vigouroux C, et al. Les lipodystrophies secondaires aux traitements antirétroviraux de l’infection par le VIH. Med Sci (Paris) 2006 ; 22 : 531–536. [Google Scholar]
8 Carr A, Cooper DA. Adverse effects of antiretroviral therapy. Lancet 2000 ; 356 : 1423–1430. [Google Scholar]
9 Ravaud C, Pare M, Azoulay S, et al. Impairment of the activin A autocrine loop by lopinavir reduces self-renewal of distinct human adipose progenitors. Sci Rep 2017 ; 7 : 2986. [Google Scholar]
10 Ravaud C, Paré M, Yao X, et al. Resveratrol and HIV-protease inhibitors control UCP1 expression through opposite effects on p38 MAPK phosphorylation in human adipocytes. J Cell Physiol 2019 ; 235 : 1184–1196.[Google Scholar]
11 Vernochet C, Azoulay S, Duval D, et al. Human immunodeficiency virus protease inhibitors accumulate into cultured human adipocytes and alter expression of adipocytokines. J Biol Chem 2005 ; 280 : 2238–2243.[Google Scholar]
12 Djedaini M, Peraldi P, Drici MD, et al. Lopinavir co-induces insulin resistance and ER stress in human adipocytes. Biochem Bioph Res Co 2009 ; 386 : 96–100. [Google Scholar]
13 Hilgenfeld R. From SARS to MERS: crystallographic studies on coronaviral proteases enable antiviral drug design. FEBS J 2014 ; 281 : 4085–4096. [Google Scholar]
14 Zhang L, Lin D, Sun X, et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved a-ketoamide inhibitors. Science 2020; 368 : 409–12. [Google Scholar]
15 Anand K, Ziebuhr J, Wadhwani P, et al. Coronavirus main proteinase (3CLpro) structure: basis for design of anti-SARS drugs. Science 2003 ; 300 : 1763–1767. [Google Scholar]
16 Cao B, Wang Y, Wen D, et al. A trial of lopinavir-ritonavir in adults hospitalized with severe Covid-19. N Engl J Med 2020; 382 : 1787–99. [Google Scholar]
17 Lapatto R, Blundell T, Hemmings A, et al. X-ray analysis of HIV-1 proteinase at 2.7 Å resolution confirms structural homology among retroviral enzymes. Nature 1989 ; 342 : 299–302. [Google Scholar]
18 Juckel D, Dubuisson J, Belouzard S. Les coronavirus, ennemis incertains. Med Sci (Paris) 2020, 36: 633–41. [EDP Sciences] [PubMed] [Google Scholar]u ici




